WnioskowanieStatystyczne/Elementy statystyki wielowymiarowej: Różnice pomiędzy wersjami
| (Nie pokazano 69 pośrednich wersji utworzonych przez tego samego użytkownika) | |||
| Linia 38: | Linia 38: | ||
niezależnych korelacja wynosi <math>0</math>. | niezależnych korelacja wynosi <math>0</math>. | ||
| − | ==Macierz kowariancji== | + | |
| + | |||
| + | ===Macierz kowariancji=== | ||
| Linia 47: | Linia 49: | ||
| − | dla <math>x=(x_{1,}x_{2})</math> i <math>\mu =(\mu _{1}, \mu _{2})</math> | + | dla dwóch wymiarów: <math>x=(x_{1,}x_{2})</math> i <math>\mu =(\mu _{1}, \mu _{2})</math> |
| Linia 76: | Linia 78: | ||
\end{matrix} | \end{matrix} | ||
\right] | \right] | ||
| + | </math> | ||
| + | |||
| + | |||
| + | ogólnie | ||
| + | |||
| + | |||
| + | <math> | ||
| + | S=\left[ | ||
| + | \begin{matrix} | ||
| + | \upsilon ar(x_{1}) & co\upsilon (x_{1},x_{2}) & ... & co\upsilon | ||
| + | (x_{1},x_{k}) \\ | ||
| + | co\upsilon (x_{2},x_{1}) & \upsilon ar(x_{2}) & ... & co\upsilon | ||
| + | (x_{2},x_{k}) \\ | ||
| + | ... & ... & ... & ... \\ | ||
| + | co\upsilon \left( x_{k},x_{1}\right) & co\upsilon (x_{k},x_{2}) & ... & | ||
| + | \upsilon ar(x_{k}) | ||
\end{matrix} | \end{matrix} | ||
| + | \right] | ||
</math> | </math> | ||
| − | |||
| − | == | + | <!-- |
| + | ==Dwuwymiarowy rozkład normalny== | ||
| + | |||
<math> | <math> | ||
f(t)=ke^{-\frac{1}{2}(t-\mu )A(t-\mu )^{T}} | f(t)=ke^{-\frac{1}{2}(t-\mu )A(t-\mu )^{T}} | ||
| Linia 94: | Linia 114: | ||
<math>\mu =(\mu _{1},\mu _{2})-</math> wektor wartości oczekiwanych | <math>\mu =(\mu _{1},\mu _{2})-</math> wektor wartości oczekiwanych | ||
| − | <math>k-</math> stała | + | <math>k-</math> stała normalizująca |
<math>A-</math> odwrotność macierzy kowariancji <math>C</math> | <math>A-</math> odwrotność macierzy kowariancji <math>C</math> | ||
| Linia 113: | Linia 133: | ||
</math> | </math> | ||
| + | --> | ||
| + | ==Analiza wariancji wielu zmiennych (''Multivariate ANalysis of VAriance — MANOVA'')== | ||
| − | + | Wcześniej rozpatrywaliśmy podział na grupy pomiarów opisanych jedną zmienną ([[WnioskowanieStatystyczne/Analiza_wariancji|analiza wariancji jednej zmiennej]]). Jeśli zmienna losowa <math>X</math> jest opisana wektorem | |
| + | (<math>x_{1},...,x_{k})</math>, wartość | ||
| + | wektorem o tym samym wymiarze: (<math> \mu _{1},...,\mu _{k})</math>, to | ||
| + | w miejsce wariancji mamy do czynienia z macierzą kowariancji: | ||
| − | |||
| − | |||
| − | |||
| − | |||
<math> | <math> | ||
| Linia 135: | Linia 156: | ||
\right] | \right] | ||
</math> | </math> | ||
| + | |||
gdzie: | gdzie: | ||
| + | |||
<math> | <math> | ||
\upsilon ar(x_{i})=\sigma _{x_{i}}=E((x_{i}-\mu _{i})^{2}) | \upsilon ar(x_{i})=\sigma _{x_{i}}=E((x_{i}-\mu _{i})^{2}) | ||
</math> | </math> | ||
| + | |||
<math> | <math> | ||
| Linia 146: | Linia 170: | ||
_{k})) | _{k})) | ||
</math> | </math> | ||
| + | |||
| + | |||
Zakladamy, że dane pochodzą z wielowymiarowego rozkładu normalnego, opisanego macierzą kowariancji <math>S</math> | Zakladamy, że dane pochodzą z wielowymiarowego rozkładu normalnego, opisanego macierzą kowariancji <math>S</math> | ||
| + | |||
<math> | <math> | ||
| Linia 153: | Linia 180: | ||
)^{\prime }S^{-1}(X-\mu )}{2}} | )^{\prime }S^{-1}(X-\mu )}{2}} | ||
</math> | </math> | ||
| + | |||
Jeśli <math>X</math> pochodzą z próby | Jeśli <math>X</math> pochodzą z próby | ||
| Linia 164: | Linia 192: | ||
</math> | </math> | ||
| − | Jako statystykę testową możemy | + | Jako statystykę testową możemy wybrać np. iloraz wyznaczników |
macierzy <math>S_{wew}</math> i <math>S</math>, który podlega rozkładowi | macierzy <math>S_{wew}</math> i <math>S</math>, który podlega rozkładowi | ||
<math>\Lambda </math> Wilksa: | <math>\Lambda </math> Wilksa: | ||
| + | |||
<math> | <math> | ||
| Linia 172: | Linia 201: | ||
</math> | </math> | ||
| + | =Uczenie (maszynowe, ML) nienadzorowane (unsupervised learning)= | ||
==Analiza składowych głównych (''Principal Components Analysis, PCA'')== | ==Analiza składowych głównych (''Principal Components Analysis, PCA'')== | ||
| + | Jeśli zmienne są bardzo silnie skorelowane, mamy do czynienia z redundancją. Możemy ją zmniejszyć, redukując wymiar danych. W tym celu macierz kowariancji | ||
| + | |||
| + | |||
| + | <math> | ||
| + | S=\left[ | ||
| + | \begin{matrix} | ||
| + | \upsilon ar(x_{1}) & co\upsilon (x_{1},x_{2}) & ... & co\upsilon | ||
| + | (x_{1},x_{k}) \\ | ||
| + | co\upsilon (x_{2},x_{1}) & \upsilon ar(x_{2}) & ... & co\upsilon | ||
| + | (x_{2},x_{k}) \\ | ||
| + | ... & ... & ... & ... \\ | ||
| + | co\upsilon \left( x_{k},x_{1}\right) & co\upsilon (x_{k},x_{2}) & ... & | ||
| + | \upsilon ar(x_{k}) | ||
| + | \end{matrix} | ||
| + | \right] | ||
| + | </math> | ||
| + | |||
| + | |||
| + | przedstawiamy w postaci diagonalnej | ||
| − | |||
<math> | <math> | ||
| Linia 203: | Linia 251: | ||
\right] | \right] | ||
</math> | </math> | ||
| + | |||
Wielkości <math>\lambda _{i}</math> są rozwiązaniami równania | Wielkości <math>\lambda _{i}</math> są rozwiązaniami równania | ||
<math>\left| S-\lambda I\right| =0,</math> a wektor <math>r_{i}</math> | <math>\left| S-\lambda I\right| =0,</math> a wektor <math>r_{i}</math> | ||
| − | osiami nowego układu współrzędnych. Składowe | + | osiami nowego układu współrzędnych. Składowe PCA są |
liniowymi kombinacjami obserwowanych zmiennych. | liniowymi kombinacjami obserwowanych zmiennych. | ||
| − | |||
| − | |||
| − | + | [[Plik:pca.png|center|thumb|600px|Kierunki składowych głównych (PCA) w dwóch wymiarach]] | |
| − | |||
| − | |||
| − | |||
| − | |||
| − | + | ==Analiza skupień — ''Cluster Analysis'' == | |
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | + | Wejściem dla tej klasy procedury są nieoznaczone dane, a wyjściem ich podział | |
| + | na grupy. Obecnie metody tego typu określamy czasem jako "unsupervised learning". | ||
| − | + | <!-- Można go zrealizować na wiele sposobów: <math>N</math> | |
| − | |||
| − | < | ||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
punktów <math>x^{1}...x^{N},</math> z których każdy opisany jest przez | punktów <math>x^{1}...x^{N},</math> z których każdy opisany jest przez | ||
| − | <math>k</math> cech <math>x_{1}...x_{k}</math>. | + | <math>k</math> cech <math>x_{1}...x_{k}</math>. --> |
===Metody polegające na kolejnym łączeniu punktów=== | ===Metody polegające na kolejnym łączeniu punktów=== | ||
| Linia 270: | Linia 286: | ||
Odległości między klastrami: | Odległości między klastrami: | ||
| − | * Najbliższego sąsiada ''(single linkage)'' - odległość między dwoma najbliższymi elementami klastrów | + | * Najbliższego sąsiada ''(single linkage)'' - odległość między dwoma najbliższymi elementami klastrów ''A'' i ''B'': <math>d(A,B)=\min_{x,y}d(x,y),\ \ \ \ \ x\in A,\ y\in B</math> |
| − | *(''complete linkage'' ) - odległość między dwoma najbliższymi elementami klastrów | + | *(''complete linkage'' ) - odległość między dwoma najbliższymi elementami klastrów ''A'' i ''B'': <math>d(A,B)=\max_{x,y}d(x,y),\ \ \ \ \ x\in A,\ y\in B </math> |
*''(centroid)'' - odległość między środkami klastrów, | *''(centroid)'' - odległość między środkami klastrów, | ||
| − | *''( | + | *''(average)'' - średnia odległości, itd... |
| + | |||
===Metoda K–średnich (''K – means'' )=== | ===Metoda K–średnich (''K – means'' )=== | ||
| Linia 279: | Linia 296: | ||
Wybieramy ilość klastrów, podział dokonywany jest w iteracyjnej | Wybieramy ilość klastrów, podział dokonywany jest w iteracyjnej | ||
procedurze dążącej do minimalizacji stosunku wariancji pomiędzy | procedurze dążącej do minimalizacji stosunku wariancji pomiędzy | ||
| − | klastrami do wariancji wewnątrz klastrów | + | klastrami do wariancji wewnątrz klastrów — niejako <math>ANOVA</math> |
bez ustalonego wstępnie przyporządkowania, maksimum <math> F</math> | bez ustalonego wstępnie przyporządkowania, maksimum <math> F</math> | ||
poszukiwane drogą przemieszczania elementów między klastrami. | poszukiwane drogą przemieszczania elementów między klastrami. | ||
Wersja z 18:25, 1 cze 2023
Wnioskowanie_Statystyczne_-_wykład
Spis treści
Elementy statystyki wielowymiarowej
Przypomnijmy najpierw pojęcia
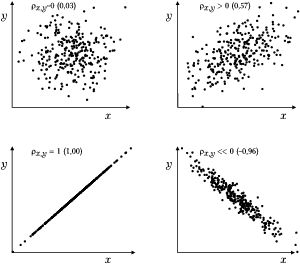
Kowariancja i współczynnik korelacji
Miarą związku między zmiennymi [math]x[/math] i [math]y[/math] jest kowariancja
[math] \sigma_{x, y} = E\left( (x-\mu_{x})(y-\mu_{y})\right) [/math]
lub unormowany do jedności współczynnik korelacji zmiennych [math]x[/math] i [math]y[/math]:
[math] \rho_{x, y}= \frac{\sigma_{x, y}}{\sigma_x \sigma_y}= \frac{E\left( \left(x-\mu_{x})(y-\mu_{y}\right)\right)} {\sqrt{E\left( (x-\mu_{x})^2\right) E\left( (y-\mu_{y})^2\right)}}, [/math]
gdzie [math]\mu_x[/math] i [math]\mu_y[/math] to odpowiednio wartości oczekiwane zmiennych [math]x[/math] i [math]y[/math]. Jeśli zmienne [math]x[/math] i [math]y[/math] związane są deterministyczną zależnością liniową (typu [math]y=c_1 x+c_2[/math]), to ich korelacja wynosi [math]1[/math] (lub [math]-1[/math], jeśli [math]c_1\lt 0[/math]). Jeśli wzrostowi zmiennej [math]x[/math] towarzyszy statystycznie wzrost zmiennej [math]y[/math], to ich korelacja jest dodatnia (pomiędzy [math]0[/math] a [math]1[/math]). Dla zmiennych niezależnych korelacja wynosi [math]0[/math].
Macierz kowariancji
[math] C=E[(x-\mu )(x-\mu )^{T}], \qquad \\ c_{ij}=E[(x_{i}-\mu_{i})(x_{j}-\mu _{j})] [/math]
dla dwóch wymiarów: [math]x=(x_{1,}x_{2})[/math] i [math]\mu =(\mu _{1}, \mu _{2})[/math]
[math] C=E\left[ \left(\begin{matrix}{x_{1}-\mu _{1}}\\ {x_{2}-\mu _{2}} \end{matrix} \right) \left( x_{1}-\mu_{1},x_{2}-\mu _{2}\right) \right] = [/math]
[math] \left[ \begin{matrix} E\left[ \left( x_{1}-\mu _{1}\right) ^{2}\right] & E\left[ \left( x_{1}-\mu _{1}\right) \left( x_{2}-\mu _{2}\right) \right] \\ E\left[ \left( x_{2}-\mu _{2}\right) \left( x_{1}-\mu _{1}\right) \right] & E \left[ \left( x_{2}-\mu _{2}\right) ^{2}\right] \end{matrix} \right] = [/math]
[math]
\left[
\begin{matrix}
\sigma _{1}^{2} & \sigma _{12} \\
\sigma _{21} & \sigma _{2}^{2}
\end{matrix}
\right]
[/math]
ogólnie
[math]
S=\left[
\begin{matrix}
\upsilon ar(x_{1}) & co\upsilon (x_{1},x_{2}) & ... & co\upsilon
(x_{1},x_{k}) \\
co\upsilon (x_{2},x_{1}) & \upsilon ar(x_{2}) & ... & co\upsilon
(x_{2},x_{k}) \\
... & ... & ... & ... \\
co\upsilon \left( x_{k},x_{1}\right) & co\upsilon (x_{k},x_{2}) & ... &
\upsilon ar(x_{k})
\end{matrix}
\right]
[/math]
Analiza wariancji wielu zmiennych (Multivariate ANalysis of VAriance — MANOVA)
Wcześniej rozpatrywaliśmy podział na grupy pomiarów opisanych jedną zmienną (analiza wariancji jednej zmiennej). Jeśli zmienna losowa [math]X[/math] jest opisana wektorem ([math]x_{1},...,x_{k})[/math], wartość wektorem o tym samym wymiarze: ([math] \mu _{1},...,\mu _{k})[/math], to w miejsce wariancji mamy do czynienia z macierzą kowariancji:
[math]
S=\left[
\begin{matrix}
\upsilon ar(x_{1}) & co\upsilon (x_{1},x_{2}) & ... & co\upsilon
(x_{1},x_{k}) \\
co\upsilon (x_{2},x_{1}) & \upsilon ar(x_{2}) & ... & co\upsilon
(x_{2},x_{k}) \\
... & ... & ... & ... \\
co\upsilon \left( x_{k},x_{1}\right) & co\upsilon (x_{k},x_{2}) & ... &
\upsilon ar(x_{k})
\end{matrix}
\right]
[/math]
gdzie:
[math]
\upsilon ar(x_{i})=\sigma _{x_{i}}=E((x_{i}-\mu _{i})^{2})
[/math]
[math]
co\upsilon (x_{i},x_{k})=\sigma _{x_{i},x_{k}}=E((x_{i}-\mu _{i})(x_{k}-\mu
_{k}))
[/math]
Zakladamy, że dane pochodzą z wielowymiarowego rozkładu normalnego, opisanego macierzą kowariancji [math]S[/math]
[math]
\Phi (X)=\frac{1}{\sqrt{(2\pi )^{k}}\sqrt{\left| S\right| }}e^{-\frac{(X-\mu
)^{\prime }S^{-1}(X-\mu )}{2}}
[/math]
Jeśli [math]X[/math] pochodzą z próby
podzielonej na grupy, to podobnie jak w ANOVA możemy
skonstruować macierze wariancji wewnątrzgrupowych i międzygrupowych i
dowieść, że [math]S=S_{wew}+S_{pom}[/math].
Testujemy hipotezę o równości średnich w grupach
[math] H_0: \mu_1 = \mu_2 = \cdots = \mu_k [/math]
Jako statystykę testową możemy wybrać np. iloraz wyznaczników macierzy [math]S_{wew}[/math] i [math]S[/math], który podlega rozkładowi [math]\Lambda [/math] Wilksa:
[math]
\Lambda =\frac{\left| S_{wew}\right| }{\left| S\right| }=\frac{\left| S_{wew}\right| }{\left| S_{wew}+S_{pom}\right| }
[/math]
Uczenie (maszynowe, ML) nienadzorowane (unsupervised learning)
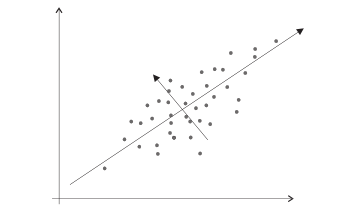
Analiza składowych głównych (Principal Components Analysis, PCA)
Jeśli zmienne są bardzo silnie skorelowane, mamy do czynienia z redundancją. Możemy ją zmniejszyć, redukując wymiar danych. W tym celu macierz kowariancji
[math]
S=\left[
\begin{matrix}
\upsilon ar(x_{1}) & co\upsilon (x_{1},x_{2}) & ... & co\upsilon
(x_{1},x_{k}) \\
co\upsilon (x_{2},x_{1}) & \upsilon ar(x_{2}) & ... & co\upsilon
(x_{2},x_{k}) \\
... & ... & ... & ... \\
co\upsilon \left( x_{k},x_{1}\right) & co\upsilon (x_{k},x_{2}) & ... &
\upsilon ar(x_{k})
\end{matrix}
\right]
[/math]
przedstawiamy w postaci diagonalnej
[math]
S=\left[
\begin{matrix}
r_{11} & r_{12} & ... & r_{1k} \\
r_{21} & r_{22} & ... & r_{2k} \\
... & ... & ... & ... \\
r_{k1} & r_{k2} & ... & r_{kk}
\end{matrix}
\right] \left[
\begin{matrix}
\lambda _{1} & 0 & ... & 0 \\
0 & \lambda _{2} & ... & 0 \\
... & ... & ... & ... \\
0 & 0 & ... & \lambda _{k}
\end{matrix}
\right] \left[
\begin{matrix}
r_{11} & r_{21} & ... & r_{k1} \\
r_{12} & r_{22} & ... & r_{k2} \\
... & ... & ... & ... \\
r_{1k} & r_{2k} & ... & r_{kk}
\end{matrix}
\right]
[/math]
Wielkości [math]\lambda _{i}[/math] są rozwiązaniami równania
[math]\left| S-\lambda I\right| =0,[/math] a wektor [math]r_{i}[/math]
osiami nowego układu współrzędnych. Składowe PCA są
liniowymi kombinacjami obserwowanych zmiennych.
Analiza skupień — Cluster Analysis
Wejściem dla tej klasy procedury są nieoznaczone dane, a wyjściem ich podział na grupy. Obecnie metody tego typu określamy czasem jako "unsupervised learning".
Metody polegające na kolejnym łączeniu punktów
Startujemy z N klastrów jednopunktowych, w każdym kroku łączymy najbliższe. Wynikiem działania jest drzewo łączenia, na którym sami musimy wybrać ilość klastrów. Wynik zależy silnie od przyjętych definicji odległości między klastrami oraz definicji odległości między punktami.
Odległości między punktami:
- Odległość Euklidesowa [math]d(x,y)=\sqrt{\underset{i=1}{\overset{k}{\sum }}(x_{i}-y_{i})^{2}}[/math] (czuła na różne skale cech).
- Odległość korelacyjna [math]d(x,y)=1-\rho (x,y),[/math] gdzie [math]\rho (x,y)=\frac{\underset{i=1}{\overset{k}{\sum }}(x-\overline{x})(y- \overline{y})}{\sigma _{x}\sigma _{y}} [/math] (znormalizowana do przedziału (0,2), mniejsza im lepiej skorelowane punkty).
Odległości między klastrami:
- Najbliższego sąsiada (single linkage) - odległość między dwoma najbliższymi elementami klastrów A i B: [math]d(A,B)=\min_{x,y}d(x,y),\ \ \ \ \ x\in A,\ y\in B[/math]
- (complete linkage ) - odległość między dwoma najbliższymi elementami klastrów A i B: [math]d(A,B)=\max_{x,y}d(x,y),\ \ \ \ \ x\in A,\ y\in B [/math]
- (centroid) - odległość między środkami klastrów,
- (average) - średnia odległości, itd...
Metoda K–średnich (K – means )
Wybieramy ilość klastrów, podział dokonywany jest w iteracyjnej procedurze dążącej do minimalizacji stosunku wariancji pomiędzy klastrami do wariancji wewnątrz klastrów — niejako [math]ANOVA[/math] bez ustalonego wstępnie przyporządkowania, maksimum [math] F[/math] poszukiwane drogą przemieszczania elementów między klastrami.